https://arxiv.org/abs/2410.11910
Explainable AI Methods for Multi-Omics Analysis: A Survey
Advancements in high-throughput technologies have led to a shift from traditional hypothesis-driven methodologies to data-driven approaches. Multi-omics refers to the integrative analysis of data derived from multiple 'omes', such as genomics, proteomics,
arxiv.org
explainable는 딱히 필요 없어서...



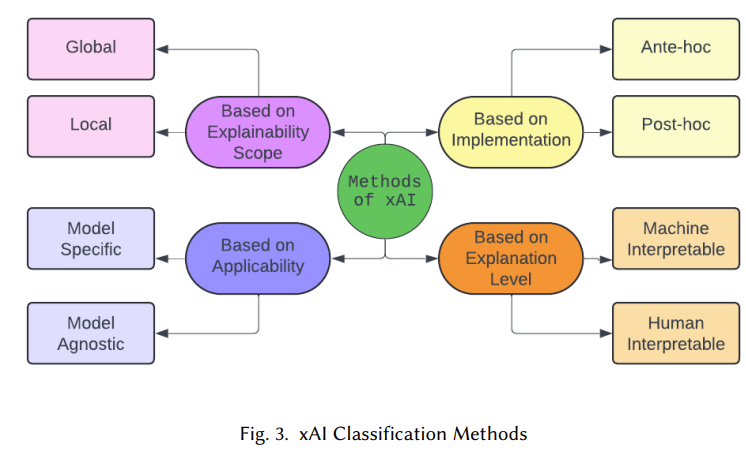
| 연구 배경 | - Multi-Omics는 유전체, 전사체, 단백체 등 다양한 생물학적 층을 통합해 질병 이해도 및 진단 정확도 향상 - 딥러닝이 성능 면에서 우수하지만, 해석력 부족으로 인해 임상 적용에 어려움 존재 (Black-box 문제) |
| 연구 목적 | - Multi-omics 분석에 딥러닝을 적용하면서 생기는 불투명성을 해소하기 위해 설명 가능한 인공지능(xAI) 기법들을 체계적으로 분석 및 분류 |
| xAI 분류 기준 | ① 범위(Scope): 전역(Global) vs 지역(Local) ② 구현 시점: 사전(Ante-hoc) vs 사후(Post-hoc) ③ 모델 특이성: 특정 모델 기반 vs 범용(model-agnostic) ④ 해석 수준: 기계해석 vs 인간해석 |
| 대표 xAI 기법 | - SHAP, LIME, PDP, ALE, PermFIT (모델 비독립) - Attention, CAM, Grad-CAM, IG, LRP, DeepLIFT, CAV, Counterfactual (모델 특화) - 시각화: Saliency Map, Heatmap, t-SNE, UMAP |
| 활용 사례 | - NSCLC, BRCA, KIRC 등 다양한 암 예측 및 서브타입 분류 - Autoencoder, CNN, Graph Attention Network 등 다양한 딥러닝 모델 기반 - TCGA, METABRIC, GDSC 등 공개 대규모 multi-omics 데이터셋 활용 |
| 장점 (기여) | ✅ 딥러닝 모델의 예측 신뢰도 및 생물학적 해석력 향상 ✅ 바이오마커 도출 및 질병 메커니즘 이해 가능 ✅ 개인맞춤 정밀의료 기반 기술로 발전 가능 |
| 주요 한계 | ❌ 샘플 수 부족 및 데이터 편향 (인구 다양성 부족) ❌ 높은 차원성과 오믹스 간 복잡한 상호작용 설명 어려움 ❌ 생물학적 해석보다는 계산적 프레임워크에 편중 ❌ 학습된 모델 간 일반화와 재현성 부족 |
| 향후 방향 | 🔬 xAI 성능-해석성 균형 향상 🌐 다양한 오믹스 간 설명 일관성 확보 👨⚕️ 의료 전문가와 비전문가 모두 이해 가능한 시각화 도구 설계 🔐 데이터 다양성과 윤리성 보장 📊 표준화된 평가 지표 및 프레임워크 개발 필요 |
| 결론 | xAI는 multi-omics 기반 정밀의료와 생명정보학 연구를 위한 핵심 도구로 부상 중이며, 향후 딥러닝의 신뢰성과 해석 가능성을 높이는 데 필수적인 기술임 |
https://arxiv.org/abs/1802.02511
DeepHeart: Semi-Supervised Sequence Learning for Cardiovascular Risk Prediction
We train and validate a semi-supervised, multi-task LSTM on 57,675 person-weeks of data from off-the-shelf wearable heart rate sensors, showing high accuracy at detecting multiple medical conditions, including diabetes (0.8451), high cholesterol (0.7441),
arxiv.org
이 것도 너무 옛날 논문...
웨어러블을 통해 예측하는 건 이미 다 했으니,...

| 연구 배경 및 문제의식 | - 웨어러블 기기(Apple Watch 등)는 방대한 심박수 데이터를 생성하지만 대부분 비라벨 데이터 - 고혈압, 당뇨, 수면무호흡증 등은 높은 유병률 대비 낮은 진단율 - 의료 라벨 수집은 비용이 크고 제한적 → 라벨 부족 문제 해결 필요 |
| 연구 목표 | 반지도 학습 기반 LSTM을 사용하여 심박수/활동량 시계열로 질환을 조기 예측 비침습적이고 접근성 높은 진단 도구로서 웨어러블 기반 헬스케어 시스템 가능성 제시 |
| 데이터셋 | - 총 14,011명 참여 (IRB 승인) - Apple Watch 기반 PPG 심박수 + 걸음 수 - 총 57,675 person-weeks - 라벨: 고혈압, 고콜레스테롤, 수면무호흡, 당뇨 |
| 모델 구조 | - 입력: multi-channel 시계열 (심박, 걸음수, dt) - CNN (3층) → BiLSTM (4층) → 1D conv output - Multi-task 출력 (4질환 동시 예측) - Dropout, max-pooling 등 regularization 적용 |
| 학습 방식 | ✅ Supervised learning ✅ Heuristic Pretraining (HRV-derived feature 예측으로 초기화) ✅ Unsupervised Sequence Learning (autoencoder로 pretrain) |
| 실험 결과 (AUC) | |
| LSTM (no pretrain) | 0.8451 |
| Heuristic Pretraining | 0.8366 |
| Sequence Pretraining | 0.7998 |
| 기존 최고 ML (LR, SVM 등) | ~0.79 |
| 핵심 인사이트 | - Semi-supervised learning → 라벨 효율성 10배 향상 - Sequence pretraining은 특히 수면무호흡·고혈압에서 효과적 - 심박 반응 (HRV)의 non-linear feature 학습이 rule-based feature보다 우수 |
| 한계점 | - 약물 등 confounding 요인 - 의료 배포 어려움 (EMR 통합, 책임소재) - 모델 해석력 부족 (black-box LSTM) - 긴 시계열 모델링 어려움 (Phased LSTM 등 필요) |
| 기여 및 의의 | - 웨어러블 데이터 기반 질환 조기 예측 모델 최초 제안 - 의료 도메인에 반지도 학습 적용하여 데이터 효율성 향상 - Multi-task LSTM 구조로 다질환 예측 성공 - 공공보건 선별 도구로서의 실용 가능성 입증 |
https://pmc.ncbi.nlm.nih.gov/articles/PMC8366414/
Cox-sMBPLS: An Algorithm for Disease Survival Prediction and Multi-Omics Module Discovery Incorporating Cis-Regulatory Quantitat
Abstract Background The development of high-throughput techniques has enabled profiling a large number of biomolecules across a number of molecular compartments. The challenge then becomes to integrate such multimodal Omics data to gain insights into biolo
pmc.ncbi.nlm.nih.gov

| 단계 | 설명 |
| A. Multi-Omics Data 구성 | - 입력 데이터는 세 가지 오믹스 데이터 블록으로 구성됨: ① mRNA expression X^(1), ② Genotypes X^(2), ③ DNA methylation X^(3) - 각 행은 sample (예: 환자), 각 열은 feature (유전자, SNP, CpG 사이트)를 나타냄 - 왼쪽의 벡터 는 각 sample의 생존 시간(time to event)을 의미함 |
| B. Cis-QTL 기반 블록 분할 | - 각 Omics 블록을 cis-regulatory 정보 (eQTL, meQTL, eQTM)에 따라 분할 예: ▪ X^(1): eQTL 유전자를 포함한 부분과 그 외(non-eQTL) ▪ X^(2): meQTL-SNP와 non-meQTL-SNP ▪ X^(3): eQTM-CpG와 non-eQTM CpG |
| C. 잔차 처리 및 생존 시간 재가중 | - 각 QTL 쌍에 대해 중복되는 생물학적 정보를 제거하기 위해 다음을 수행: ▪ eQTL: SNP의 영향을 제거한 유전자 residual u_j^{eQTL} 생성 ▪ meQTL: CpG의 영향을 제거한 SNP residual u_j^{meQTL} 생성 ▪ eQTM: 유전자의 영향을 제거한 CpG residual u_j^{eQTM} 생성 - 생존 시간 y는 censoring을 고려하여 inverse probability weighting으로 보정된 y^*로 대체 |
| D. Cox-sMBPLS 모델 학습 및 모듈 도출 | - 업데이트된 데이터들과 보정된 생존 시간을 사용하여 Supervised Cox-sMBPLS 알고리즘 수행 ▪ 각 Omics 블록에서 latent component 추출 ▪ 이들을 통합하여 생존 예측 모델 학습 - 결과로서 다중 오믹스 모듈 (Multi-omics Modules)이 도출됨 → 하나의 모듈은 특정 gene, SNP, CpG 조합으로 구성되어 있고, 특정 생존 특성과 높은 관련을 가짐 |
| 핵심 요소 | 내용 |
| 입력 | 생존 시간 y, mRNA, Genotype, Methylation 데이터 |
| 중간 처리 | cis-QTL 정보에 기반한 잔차 생성, 생존 시간 censoring 보정 |
| 모델 | sparse Multi-block PLS 기반의 Cox 모델 (Cox-sMBPLS) |
| 결과 | 생존 예측 정확도 향상 + 해석 가능한 multi-omics module 도출 |

| 패널 | 내용 |
| A. Gene-set Network | - 왼쪽 원: module 13에 포함된 유전자 간의 co-expression network (연결 강도에 따라 선 색상 표시) → 전체 유전자 간 82%가 co-expressed 되어 있음 - 오른쪽 원: module 13과 10의 차이 유전자들만 따로 분리하여 네트워크 분석 → 이들 간 100% co-expression → 생물학적으로 매우 강한 연결성을 지님 ▪ 이 차이가 모듈 13이 더 유의미해졌던 원인임 |
| B. Gene-Disease Network | - 네 개의 핵심 유전자 PDPK1, TAB2, PRICKLE3, HRC와 관련된 질병 맵 → 심부전(Heart Failure), 심근 비대(Cardiac Hypertrophy), 심장 기형 등과 강하게 연결됨 → 중심 유전자들이 cardio-related 질환과 다중적으로 연관되어 있음 |
| C. Disease Enrichment Table | - module 13에 포함된 유전자 기반으로 DisGeNET을 통해 질병 연관성 분석 → 모든 질환이 FDR<0.05로 유의미 → 특히 Heart Failure (모든 형태 포함), Cardiomegaly, Cardiac Hypertrophy, Myocardial Failure 등 심장질환 관련 항목이 두드러짐 → Bgene Ratio는 해당 질환과 연관된 유전자의 비율을 의미 |
| D. Multi-Omics Chromosomal Map | - module 13에서 도출된 유전자, SNP, CpG의 염색체상 위치 시각화 ▪ 파란색: 유전자 ▪ 빨간색: SNP ▪ 노란색: CpG - 노란 박스로 묶인 부분은 동일한 염색체 상의 좁은 영역에 2개 이상 오믹스 요소가 존재하는 경우 → 다중 오믹스 요소가 같은 영역에서 집적되어 있음은 생물학적으로 중요한 hotspot 가능성을 의미 → 예: Chr1, Chr3, Chr6, Chr7 등 |
| 항목 | 설명 |
| 모듈 13의 타당성 | module 10보다 더 낮은 p-value (0.059 vs 0.097)를 가지며, 공통되지 않은 유전자들이 100% co-expression을 보여 기능적 결속력이 강함 |
| 중심 유전자 | PDPK1, TAB2, PRICKLE3, HRC는 모두 심장 질환 또는 심부전과 관련된 경로에 연관 |
| 질병 연관성 | Disease Enrichment에서 다양한 심장 질환과 유의한 연관성이 나타남 |
| 염색체 기반 통합성 | SNP, 유전자, CpG가 동일 위치에 클러스터링 되어 있어 multi-omics 통합의 생물학적 설득력을 제공 |
이게 뭔 말인지....
일단 간단하게 조사만 하고 나중에 쓰인다면 집중해서 읽어볼 것 같네요...
| 연구 목적 | 고차원 멀티-오믹스 데이터를 통합하여 생존 시간(time-to-event)을 예측하고, 생물학적으로 유의미한 feature 모듈을 식별하기 위한 통합 알고리즘 개발 |
| 핵심 아이디어 | 🔹 Multi-block Sparse PLS + Cox-PH 모델 통합 🔹 cis-regulatory 정보 (eQTL, meQTL, eQTM)를 활용한 Omics 블록 간 상호작용 반영 🔹 Inverse probability weighting으로 censoring 보정된 생존시간 사용 |
| 모델 이름 | Cox-sMBPLS (Supervised Cox sparse Multi-Block Partial Least Squares) |
| 입력 데이터 | - mRNA expression (27,645 genes) - SNPs (578,846 variants) - DNA methylation (12,283 CpGs) - 생존 시간 + censoring indicator |
| 데이터 출처 | 심부전 환자 91명 (UIC 병원 cohort) + 외부 QTL reference (GTEx, BIOS QTL) |
| 방법론 구성 | ① 생존시간 censoring 보정 → y^* ② QTL 정보 기반 feature 분리 및 잔차 생성 ③ 블록별 latent component 추출 (sparse PLS) ④ 전체 latent vector 결합 후 Cox-PH 모델 학습 ⑤ multi-omics module 도출 |
| 비교 모델 | Elastic Net Cox, Random Survival Forest (RSF), Block Forest, MCIA |
| 예측 성능 결과 | ✅ Cox-sMBPLS가 전 범위에서 가장 높은 C-index (0.60~0.64) ✅ censoring이 60%로 높아져도 성능 하락폭 최소화 (2~4%) |
| 실제 데이터 분석 | - 15개의 multi-omics module 도출 - Module 13: 유의미한 생존 예측 (p = 0.059), 497개 feature 포함 - 핵심 유전자: PDPK1, TAB2, HRC, PRICKLE3 (심장질환 관련) |
| 생물학적 해석 | - Gene co-expression: 100% (module 13) - Disease enrichment: 심부전, 심근비대 등 유의미하게 연관 - 염색체 지도에서 SNP-gene-CpG가 같은 위치에 클러스터링 |
| 한계점 | - pathway나 PPI network 정보 미포함 - 기능적 생물학 실험(in vivo validation)은 수행되지 않음 - 계산 복잡도 높음 |
| 기여 | ✅ 생존 분석 + 오믹스 통합 + biological prior를 결합한 최초 모델 ✅ 예측 성능 + 해석 가능성 동시 확보 ✅ 유의미한 multi-omics module 도출로 바이오마커 발굴 가능성 제시 |
| 활용 가능성 | ▶ 정밀의료용 생존 예측 모델 ▶ 생존 관련 바이오마커 후보 발굴 ▶ 다중 오믹스 통합 분석 및 기능 해석 연구 |
https://pmc.ncbi.nlm.nih.gov/articles/PMC11139513/
Pathformer: a biological pathway informed transformer for disease diagnosis and prognosis using multi-omics data - PMC
Abstract Motivation Multi-omics data provide a comprehensive view of gene regulation at multiple levels, which is helpful in achieving accurate diagnosis of complex diseases like cancer. However, conventional integration methods rarely utilize prior biolog
pmc.ncbi.nlm.nih.gov

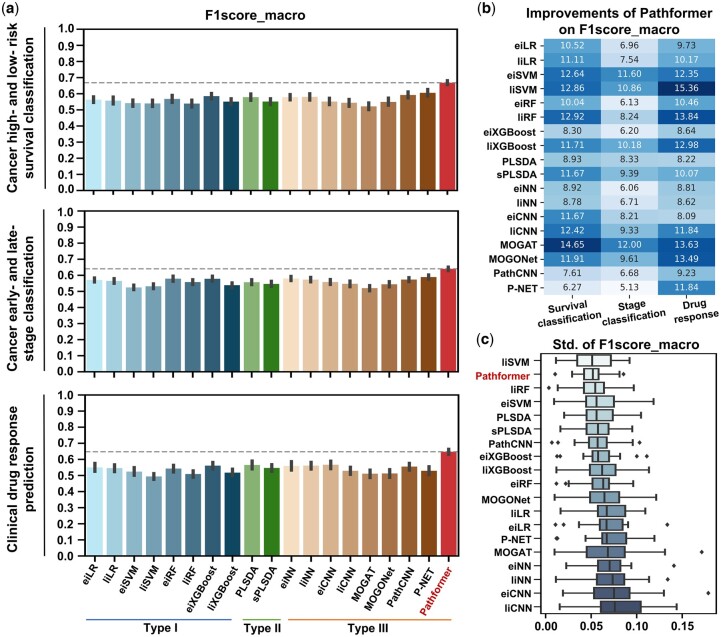

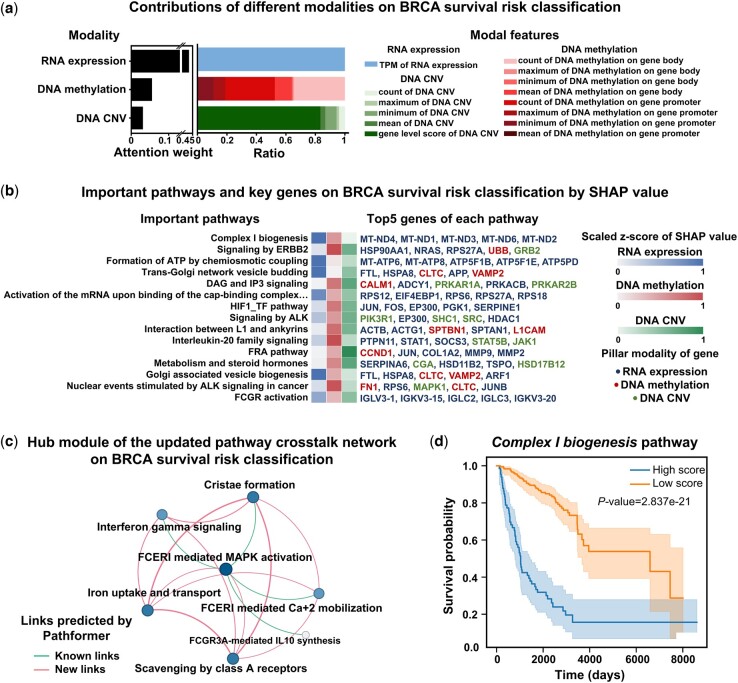
| 연구 목적 | 다양한 multi-omics 데이터를 생물학적 지식에 기반해 통합하고, 질병 진단/예후/약물 반응 예측에서 높은 정확도와 해석력을 동시에 달성하는 AI 모델 제안 |
| 모델 이름 | Pathformer (Biological Pathway-informed Transformer) |
| 핵심 아이디어 | ① gene-level에서 다양한 omics 정보를 통합한 임베딩 생성 ② gene→pathway 변환을 sparse network로 수행 (PSNN) ③ pathway crosstalk network를 bias로 반영한 criss-cross attention 기반 Transformer 적용 |
| 입력 데이터 | - TCGA: RNA expression, DNA methylation, CNV (tissue) - cfRNA-seq: plasma & platelet RNA 변형 정보 포함 (liquid biopsy) → 총 3~7 modalities 사용 |
| 모듈 구성 | ① Biological Pathway + Crosstalk 생성 ② Multi-omics input ③ Biological Multi-modal Embedding (EG → EP) ④ Criss-cross Transformer (pathway ↔ omics 정보 교환) ⑤ Classifier (FCNN) ⑥ Interpretability (SHAP, Attention) |
| 비교 대상 | 총 18개 모델과 비교 (SVM, RF, CNN, GNN, P-NET, MOGONet 등) |
| 성능 결과 | - 생존 예측: 평균 F1_macro +6.3 ~ 14.6% 향상- 병기 예측: 평균 +5.1 ~ 12% 향상 - 약물 반응 예측: 평균 +8.1~13.6% 향상 - 액체 생검: 민감도 48.8% @ 99% specificity |
| 해석 가능성 | - SHAP 기반으로 주요 경로/유전자/오믹스 modality 식별 - 실제 알려진 유방암 유전자와 일치 (ex. Complex I Biogenesis, MT-ND*) - pathway crosstalk network를 업데이트하여 허브 경로 네트워크 시각화 가능 |
| 차별점 | - 기존 모델은 omics 통합 또는 경로 정보만 사용 → Pathformer는 gene→pathway 변환과 경로 간 상호작용까지 통합 모델에 학습시킴 - Criss-cross attention 구조는 기존 Transformer보다 omics-context 학습에 유리 |
| 한계점 | ① Non-coding RNA 제외됨 ② cfRNA 멀티오믹스 데이터가 부족함 ③ 경로 수 확장 시 메모리 병목 ④ SHAP 기반 해석은 생물학적 실험 필요 |
| 기여 요약 | - 경로 기반 Transformer 구조의 설계 및 구현 - Criss-cross attention + biological bias 통합 - 액체 생검 조기 암 진단 가능성 제시 - 생물학적으로 해석 가능한 AI 모델 구현 - 오픈소스 코드 및 데이터 공개 🔗GitHub |
'인공지능 > 논문 리뷰 or 진행' 카테고리의 다른 글
| Few-shot 관련 논문 (0) | 2025.05.31 |
|---|---|
| 데이터 기반 질환 예측 논문 정리 - 3 (3) | 2025.05.29 |
| 데이터 기반 질환 예측 논문 정리 - 1 (3) | 2025.05.22 |
| MAQA: Evaluating Uncertainty Quantification in LLMs Regarding Data Uncertainty (4) | 2025.05.22 |
| Adversarial Attacks in NLP 관련 논문 정리 - 6 (0) | 2025.05.19 |